5. Juni 2023 | Neuigkeiten & Hinweise:
Gemeinsame Forschung mit PeptiStar Inc. zur Unterstützung der Entwicklung von Therapeutika auf Nukleinsäurebasis
Veröffentlichung einer Software zur Oligonukleotid-Charakterisierung, LabSolutions Insight Biologics
Am 5. Juni veröffentlicht die Shimadzu Corporation LabSolutions Insight Biologics, eine Software zur Oligonukleotid-Charakterisierung für die Hochleistungsflüssigkeitschromatographen mit Quadrupol-Flugzeit-Massenspektrometern (Q-TOF) LCMS-9030 und LCMS-9050. Die Software führt Messung und Analyse bei der Oligonukleotid-Charakterisierung automatisch durch und reduziert die Analysezeit im Vergleich zu herkömmlichen Verfahren auf ein Zehntel oder weniger. Ausgestattet mit Funktionen wie der Anzeige von Molekülstrukturformeln, Vorschlägen zur Berechnung von Komponentenverhältnissen und einer zum Patent angemeldeten Technologie zur Verunreinigungsbestimmung, trägt die Software zur Effizienzsteigerung der Forschung an Nukleinsäure-basierten Therapeutika bei.
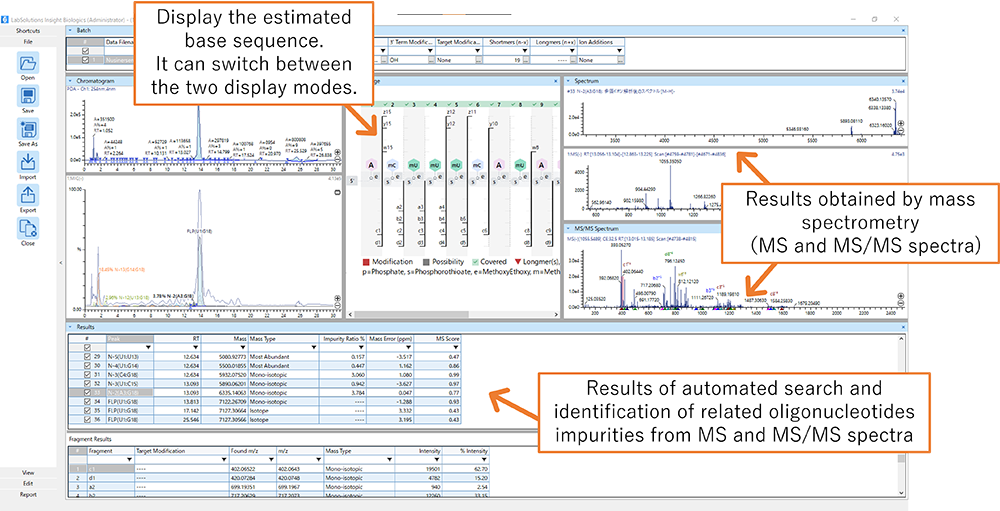
Foto: LabSolutions Insight Biologics Softwarefenster
Nukleinsäurebasierte Therapeutika sind eine Art von Biopharmazeutika, die aus DNA und RNA bestehen. Sie weisen minimale Nebenwirkungen auf und gelten als vielversprechend für die Behandlung bisher schwer behandelbarer Krankheiten. Nach niedermolekularen und Antikörper-basierten Arzneimitteln werden sie als „dritte vielversprechende Arzneimittelklasse“ angesehen. Oligonukleotide, die pharmazeutischen Wirkstoffe nukleinsäurebasierter Therapeutika, werden in der Regel chemisch synthetisiert. Dabei können jedoch unbeabsichtigte Verunreinigungen entstehen. Sicherheitsbestätigungen und -bewertungen sind erforderlich, doch da die Molekulargewichte der Hauptkomponenten ähnlich sind, sind präzise Gewichtsmessungen notwendig, was die Charakterisierung erschwert.
LabSolutions Insight Biologics wurde auf Basis der Ergebnisse einer gemeinsamen Forschungsarbeit mit PeptiStar Inc., einem Auftragsentwicklungs- und -herstellungsunternehmen (CDMO) für Peptidpharmaka *1 und Nukleinsäure-basierte Wirkstoffe, entwickelt. Zur Evaluierung des Charakterisierungsprozesses wurden Proben verwendet, die den von PeptiStar bereitgestellten Nukleinsäure-basierten Therapeutika nachempfunden waren, wodurch eine hochpräzise Charakterisierung erreicht wurde. Die Software ist benutzerfreundlich gestaltet, da sie auf dem für diese Forschung erforderlichen Arbeitsablauf basiert – von der Forschung und Entwicklung über die Herstellung der pharmazeutischen Wirkstoffe bis hin zur Reinheitsprüfung.
Merkmale
1. Charakterisierung glatter Oligonukleotide
Die Überprüfung auf Verunreinigungen, die bei der Oligonukleotidsynthese entstehen, ist zeit- und arbeitsaufwändig. Shimadzu hat mithilfe von Daten aus der gemeinsamen Forschung mit PeptiStar einen einfachen und schnellen Charakterisierungs-Workflow entwickelt. Die umfassende Suche nach den Hauptkomponenten und ihren zugehörigen Verunreinigungen erfolgt automatisch anhand der MS- und MS/MS-Spektren *2. Dadurch reduziert sich der Zeitaufwand für die Charakterisierung auf ein Zehntel oder weniger der üblicherweise benötigten Zeit.
2. Einfacher Arbeitsablauf für jeden
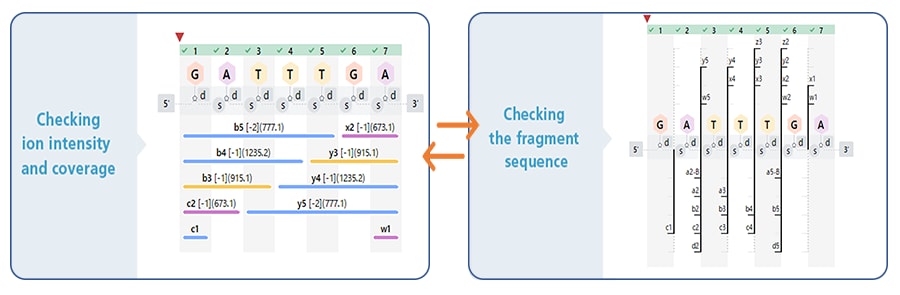
Dank unserer proprietären Softwarefunktionen ist der Arbeitsablauf für jeden Anwender einfach zu befolgen. Normalerweise muss die Nukleotidsequenz der Hauptkomponenten vor der Oligonukleotidsequenzmessung eingegeben werden – ein komplexer und fehleranfälliger Schritt. Mit einer Funktion, die die Nukleotidsequenzinformationen in Echtzeit als Strukturformel anzeigt, lassen sich die protokollierten Hauptkomponenten jedoch visuell überprüfen. Zusätzlich verfügt die Software über eine Funktion zum Umschalten der Anzeige zwischen dem vollständigen Modus (Abbildung unten links) zur Überprüfung der Genauigkeit der Basensequenz *3 und dem Verzweigungsmodus (Abbildung unten rechts) zur Überprüfung der Positionen, an denen Verunreinigungen häufig auftreten. Durch die Überprüfung der Ergebnisse in diesen beiden Modi kann die geschätzte Sequenz genauer bewertet werden.
3. Abschätzung der zur Identifizierung von Verunreinigungen erforderlichen Basensequenzen
Bei der Identifizierung von Oligonukleotidsäureanaloga ist die Suche nach Verunreinigungen mit ähnlichen Molekulargewichten und Basensequenzen eine schwierige und komplexe Aufgabe. Diese Software nutzt unsere proprietäre Fragmentspektrum-Methode (zum Patent angemeldet), um Basensequenzen selbst unter vielen ähnlichen Molekülen mit hoher Genauigkeit zu bestimmen. Die effiziente Zusammenführung mehrerer MS/MS-Spektren vereinfacht die Überprüfung der Spektralinformationen und minimiert den Einfluss von Messabweichungen.
- *1: Hierbei handelt es sich um Arzneimittel, deren Hauptbestandteile Peptide sind, die aus 2 bis 50 miteinander verbundenen Aminosäuren bestehen. Sie zeichnen sich durch ein breites Wirkungsspektrum und wenige Nebenwirkungen aus.
- *2: Diese Daten stammen aus massenspektrometrischen Analysen. Die Masse ist auf der vertikalen Achse, die Detektionsintensität auf der horizontalen Achse aufgetragen. Sie enthalten zahlreiche Informationen über die Molekulargewichte der Hauptkomponenten und Reste.
- *3: Dies sind die Einheiten für die Konfiguration von Oligonukleotiden. Sie bestehen aus vier Typen. Die Sequenz muss auf Verunreinigungen überprüft werden.
Weitere Details finden Sie unter
LabSolutions Insight Biologics


