20. Mai 2025 | News & Notices Shimadzu Corporation bringt "MicrobialTrack" auf den Markt – eine neue globale Plattform zur mikrobiellen Identifizierung, die auf der weltweit größten Datenbank basiert.
Mit der Ankündigung einer Plattform der nächsten Generation für die MALDI-TOF-MS-basierte mikrobielle Identifizierung, die die umfangreichste Datenbank mit rund 85.000 prokaryotischen Arten umfasst, darunter nicht nur bekannte, bereits beschriebene Mikroorganismen, sondern auch schwer zu kultivierende und nicht kultivierbare Mikroorganismen, definiert MicrobialTrack die Grenzen der MALDI-TOF-MS-basierten mikrobiellen Identifizierung neu.
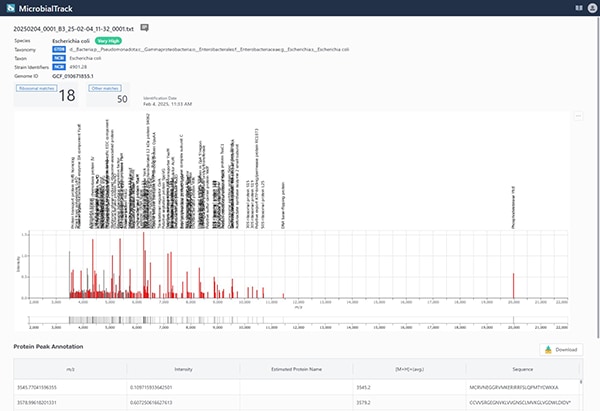
Die Shimadzu Corporation hat MicrobialTrack auf den Markt gebracht, eine innovative Softwareplattform für die hochpräzise mikrobielle Identifizierung in verschiedensten Anwendungsbereichen, darunter Lebensmittelsicherheit, Gesundheitswesen, Wirkstoffforschung und Umweltforschung. Die Software nutzt eine proprietäre, umfangreiche Proteinmassendatenbank, die auf öffentlich verfügbaren mikrobiellen Genominformationen basiert. Sie analysiert Messdaten, die mittels Matrix-unterstützter Laserdesorption/Ionisations-Flugzeitmassenspektrometrie (MALDI-TOF MS) gewonnen wurden. Die MicrobialTrack-Datenbank umfasst eine umfassende Sammlung bekannter mikrobieller Einträge mit ca. 85.000 prokaryotischen Arten (Bakterien und Archaeen), darunter solche mit gültig publizierten taxonomischen Namen sowie schwer kultivierbare und bisher nicht kultivierbare Mikroorganismen. Dies erweitert das Spektrum der Identifizierung deutlich über die Grenzen herkömmlicher Methoden hinaus.
In den letzten Jahren ist die Nachfrage nach der Identifizierung von Mikroorganismen weltweit gestiegen, bedingt durch ein erhöhtes Hygienebewusstsein, die Zunahme gekühlter Lebensmittel und das Auftreten antibiotikaresistenter Bakterien. In der klinischen und lebensmittelmikrobiologischen Diagnostik identifiziert die sogenannte „Fingerprint-Methode“ Mikroorganismen anhand von Messergebnissen der MALDI-TOF-Massenspektrometrie. Diese Methode liefert Ergebnisse in etwa drei Stunden, einschließlich der Vorbehandlung der mikrobiellen Isolate (Zellen), der MALDI-TOF-MS-Messung und der Datenanalyse. Die Datenbank ist jedoch in der Regel auf etwa 5.000 bekannte Spezies beschränkt, was die Genauigkeit der Identifizierung beeinträchtigt und die Anwendungsmöglichkeiten in vielen Bereichen der Mikrobiologie einschränkt.
Um diese Einschränkung zu überwinden, nutzt MicrobialTrack einen proteomischen Ansatz zur Identifizierung mikrobieller Arten. Dieser basiert auf der Ähnlichkeit zwischen den aus Genomen geschätzten theoretischen Proteinmassen und den in Proben nachgewiesenen Proteinmassen. Das Produkt verwendet die Datenbank für theoretische Massen prokaryotischer Mikroorganismen „GPMsDB“, die auf etwa 400.000 Genomdatensätzen aus öffentlichen Genomdatenbanken beruht. Die Analyse zielt auf ca. 85.000 prokaryotische Arten ab, die anhand genomischer Informationen vorhergesagt wurden, darunter auch schwer kultivierbare und nicht kultivierbare Mikroorganismen. Dadurch können zahlreiche Mikroorganismen identifiziert werden, die mit der Fingerprint-Methode in Kombination mit MALDI-TOF-MS nicht nachweisbar waren.
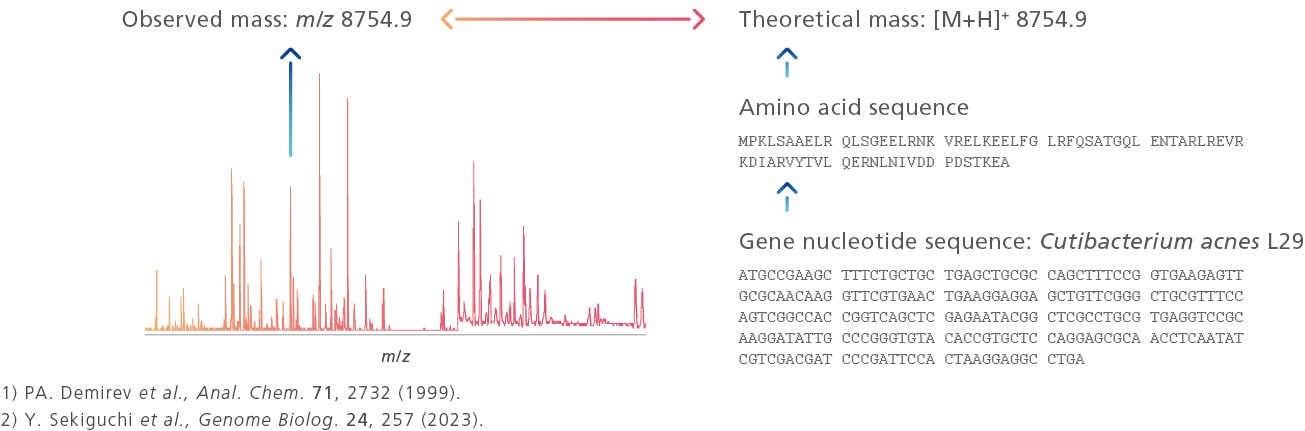
Abbildung: Prinzipien der Mikroorganismenidentifizierung mittels proteombasierter Verfahren
MicrobialTrack ist ein Cloud-basierter Dienst zur Identifizierung von Mikroorganismen im Webbrowser. Für die Nutzung ist weder ein PC noch eine Softwareinstallation erforderlich. Nutzer registrieren sich einfach und rufen anschließend die angegebene URL auf, um die Analyse zu starten. Jährlich werden mehrere hunderttausend neue prokaryotische Genomsequenzen veröffentlicht und in öffentliche Datenbanken aufgenommen. Die MicrobialTrack-Datenbank wird regelmäßig aktualisiert, um neu hinzugefügte Taxa als Genomsequenzen zu integrieren und das jeweils aktuellste, sich ständig weiterentwickelnde taxonomische System abzubilden. Nutzer haben während der gesamten Lizenzlaufzeit Zugriff auf die jeweils aktuelle MicrobialTrack-Datenbank.
MicrobialTrack ist ein Produkt, das in gemeinsamer Forschung mit dem National Institute of Advanced Industrial Science and Technology (AIST) und dem National Institute of Technology and Evaluation entwickelt wurde. Im Jahr 2023 veröffentlichten das AIST und unser Unternehmen die Grundlagen dieser Technologie zur Mikroorganismenidentifizierung, darunter die genomisch vorhergesagte Massendatenbank „GPMsDB“ und einen eigens entwickelten Algorithmus zur Analyse von Massenspektren. 1) MALDI, die Technologie, die unserem leitenden Forschungsstipendiaten Koichi Tanaka den Nobelpreis für Chemie einbrachte, beruht auf der Bestrahlung einer mit einer Matrix (Ionisationshilfsmittel) vermischten Probe mit einem Laser, um hochmolekulare Verbindungen wie Proteine zu ionisieren, ohne sie zu zerstören.
- 1) Y. Sekiguchi et al., Genome Biolog, 24 (2023).
Weitere Details finden Sie unter
Mikrobielle Spuren


